 |
特任教授 | |
| 井原 茂男 | ||
| Professor Sigeo IHARA | ||
| 所属: | 東京大学 先端科学技術研究センター システム生物医学 東京大学 大学院数理科学研究科 |
|
| 専門分野: | バイオインフォーマティクス、計算物理 | |
研究内容
統計物理、多体問題、ネットワーク、ダイナミックスの観点から生命現象を数理的にとらえる
生命科学のデータは、サンプルの数に比べて特徴を表すデータ数が多いという特徴があり、通常の統計データと大きく異なっています。 このため、従来開発されてきた方法とは異なった方法論の開発が必要になっています。 統計解析や情報理論からのアプローチからは、特徴的なデータ間での相関関係は示せるものの、遺伝子などの探索を効率的に行うには、原子や分子のレベルにたった物理学的、数学的なモデル化が必要です。 原子や分子のレベルからの第一原理的な計算からの理解する立場にたてば、多くの仮設なしに、複雑に関係しあった関係性を予測できるので大変期待されています。 計算可能な時間と空間のスケールが小さいため、階層性が複雑でしかも多数の分子が協調して生まれる生命現象を理解し、かつ予測することは現状では困難です。
そこで、既存の知識での関係性をネットワークとして表現し、関係性の特徴を調べる統計解析や情報理論だけからのアプローチだけでなく、原子や分子のレベルからの生命現象を理解するための数学や物理学的なモデル化を融合した方法論の開発と実際の実験データへの適用をめざした研究を進めています。 時間と空間での扱える範囲が広く、新しい発見ができるような数理モデルによるシミュレーションを多用した新たなマイニング方法の探索や、階層性と協調性を内在するモデル化を実験グループと共同で探っています。
生命のシステム的理解のための数理解析
刺激を与えた後の転写の過程が時間とともに進む実験結果をもとに、転写の仮定の熱力学と情報処理のモデリング、および転写を担うポリメレースの運動を輸送の数理モデルであるセルオートマトンシミュレーションから解析を進めています。DNAループ構造の変化、DNAが巻きついている蛋白質の化学的な修飾の変化などによって、転写過程がダイナミックに変化するときのメカニズムを実験家とも協力しつつ、数理モデルをたてて解析を進めています(図1)。その他、ライブセルイメージングによる具体的な結果から、細胞の遊走のダイナミカルな運動のモデリング、さらに細胞内輸送の小胞体の輸送モデルの構築も進めています。
新たなデータマイニング手法:知識処理とシミュレーションの結果の活用
最近は高速シーケンサーを用いた蛋白質とDNA(RNA)への結合データの詳細な相関関係の統計解析、その視覚化のための表示方法の開発も進めています。解析結果から意味を考えるとき、細胞内部の蛋白質がどのようにお互いに協調的に作用するのかを、蛋白質の関係性の時間的な変化をネットワークの動力学として捉え、蛋白質の関係性のつながりがどのくらいもろいのかをネットワークの堅牢性として捉える包括的な視点も大切です。この場合、蛋白質、DNA、RNAの間の変化を実験から推定し、データベース等にある既知の情報との関係を明確にしていくことになります。私たちも、文献情報やネッワークの動力学、グラフ理論的解析など様々な試みをしてきました。扱うネットワークが図として表現することが難しいスケールフリーに近いため、ネットワークの時間的な変化をどのように表現するかについても検討しています(図2)。ごく最近、ミクロな分子の構造同士の関係性を取り入れて相互作用の関係を解析をするために、既知のアミノ酸配列と分子シミュレーションの結果を用いてトポロジーの観点から蛋白質構造を分類し、蛋白質同士の結合あるいは、抗原と抗体の界面の相互作用について3次元構造の観点からの解析を始めました。
これらの研究成果をもとに更なる研究の深化を先端研の他部署、他の部局と連携して進め、産官学の連携も進めています。
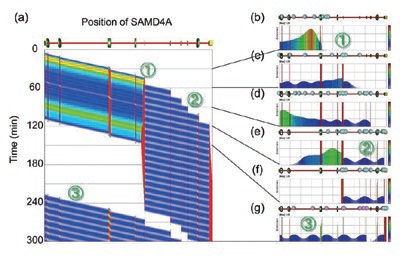 図1.転写の数理モデルから得られた遺伝子上の障害物による転写の変化の様子。
図1.転写の数理モデルから得られた遺伝子上の障害物による転写の変化の様子。
転写の数理モデルから得られた遺伝子上の障害物によって転写が変化する様子。①ポリメレースが自由に運動、②渋滞によってポリメレースの運動は強く制限される、③強く制限された運動の領域が遺伝子全体に広がる。
Ohta, Kodama, Ihara, Phys. Rev. E84 041922 (2011).
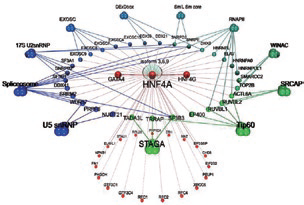 図2.時間変化するネットワークの様子。
図2.時間変化するネットワークの様子。
蛋白質(中心の赤)と他の蛋白質との相互作用ネットワークが初期(前正面)から後期(左前)まで時間的に変化する。
Daigo, Ohta et al., J. Biol.Chem., 286 674( 2011)
原著論文
バイオ関連
- Visualization of ligand-induced Gi protein activation in chemotaxing cells
K. Masuda, J. Kitakami, T. Kozasa, T. Kodama, S. Ihara and T. Hamakubo
The FASEB Journal, 2016. vol.31 no.3 910-919 doi: 10.1096/fj.201601102R - Simple model of pH-induced protein denaturation
T. Hidaka, A. Shimada, Y. Nakata, H. Kodama, H. Kurihara, T. Tokihiro, and S. Ihara
Phys. Rev. E 92, 012709 – Published 7 July 2015. doi: 10.1103/PhysRevE.92.012709 - Direct evidence for pitavastatin induced chromatin structure change in the KLF4 gene in endothelial cells,
Maejima T, Inoue T, Kanki Y, Kohro T, Guoliang Li, Ohta Y, Kimura H, Kobayashi M, Taguchi A, Tsutsumi S, Iwanari H, Yamamoto S, Aruga H, Dong S, Stevens JF, Poh HM, Yamamoto K, Kawamura T, Mimura I, Suehiro J, Sugiyama A, Kaneki K, Shibata H, Yoshinaka Y, Doi T, Asanuma A, Tanabe S, Tanaka T, Minami T, Hamakubo T, Sakai J, Nozaki N, Aburatani H, Nangaku M, Ruan X, Tanabe H, Ruan Y, Ihara S, Endo A, Kodama T, Wada Y,
PLoS One. 2014 May 05;9(5):e96005. doi: 10.1371/journal.pone.0096005 - Ultradiscrete modeling and simulation for gene transcription,
Ohta Y and Ihara S,
RIMS Kokyuroku Bessatsu, The Breadth and Depth of Nonlinear Discrete Integrable Systems, pp 101 - 124, 2013. - Path-preference cellular-automaton model for traffic flow through transit points and its application to the transcription process in human cells,
Yoshihiro Ohta, Akinobu Nishiyama, Yoichiro Wada, Yijun Ruan, Tatsuhiko Kodama, Takashi Tsuboi, Tetsuji Tokihiro, and Sigeo Ihara,
Physical Review E 86, 021918– Published 21 August 2012. doi: 10.1103/PhysRevE.86.021918 - A simple knowledge-based mining method for exploring hidden key molecules in a human biomolecular network,
Tsuji S, Ihara S, Aburatani H,
Bio Med Central Systems Biology 2012, 6:124. doi: 10.1186/1752-0509-6-124 - Epigenetically coordinated GATA2 binding is necessary for endothelium-specific endomucin expression,
Kanki Y, Kohro T, Jiang S, Tsutsumi S, Mimura I, Suehiro J, Wada Y, Ohta Y, Ihara S, Iwanari H, Naito M, Hamakubo T, Aburatani H, Kodama T, Minami T,
Published Online 10.06.2011 The EMBO Journal (2011) 30, 2582-2595. doi: 10.1038/emboj.2011.173 - Cellular-automaton model of the cooperative dynamics of RNA polymerase II during transcription in human cells,
Ohta Y, Kodama T, Ihara S,
Physical Review E 84, 041922 – Published 19 October 2011. doi: 10.1103/PhysRevE.84.041922 - Proteomic analysis of native hepatocyte nuclear factor-4α (HNF4α) isoforms, phosphorylation status, and interactive cofactors,
Daigo K, Kawamura T, Ohta Y, Ohashi R, Katayose S, Tanaka T, Aburatani H, Naito M, Kodama T, Ihara S, Hamakubo T,
The Journal of Biological Chemistry, 2011 286: 674-686. doi:10.1074/jbc.M110.154732 - Active RNA polymerases: mobile or immobile molecular machines?,
Papantonis A, Larkin JD, Wada Y. Ohta Y, Ihara S, Kodama T, and Cook P R,
PLoS Biology, 8, Issue 7, e1000419 July 13, 2010. doi:10.1371/journal.pbio.1000419 - Enhancement of bone formation ex vivo and in vivo by a helioxanthin-derivative,
Nakajima K, Komiyama Y, Hojo H, Ohba S, Yano F, Nishikawa N, Ihara S, Aburatani H, Takato T, Chung U,
Biochemical and Biophysical Research Communications, Volume 397, Issue 3, 2 July 2010, Page 631. - A Wave of nascent transcription on activated human genes,
Wada Y, Ohta Y, Xu M, Tsutsumi S, Minami T, Inoue K, Komura D, Kitakami J, Oshida N, Papantonis A, Izumi A, Kobayashi M, Meguro H, Kanki Y, Mimura I, Yamamoto K, Mataki C, Hamakubo T, Shirahige K, Aburatani H, Kimura H, Kodama T, Cook P R, Ihara S,
Proceeding of the National Academy of Sciences USA 106: (43) 18357-18361 (2009). doi: 10.1073/pnas.0902573106 - The peroxisome proliferator-activated receptor gamma/retinoid X receptor alpha heterodimer targets the histone modification enzyme PR-Set7/Setd8 gene and regulates adipogenesis through a positive feedback loop,
Wakabayashi K, Okamura M, Tsutsumi S, Nishikawa NS, Tanaka T, Sakakibara I, Kitakami J, Ihara S, Hashimoto Y, Hamakubo T, Kodama T, Aburatani H, Sakai J,
Molecular and Cellular Biology July 2009 vol. 29 no. 13 3544-3555. - Genome-wide detection of human copy number variations using high density DNA oligonuceotide arrays,
Komura D, Shen F, Ishikawa S, Fitch KR, Chen W, Liu G, Ihara S, Nakamura H, Hurles ME, Zhang J, Scherer SW, Jones KW, Shapero MH, Huang J, Lee C and Aburatani H,
Genome Research. 2006. 16: 1575-1584. - Noise reduction from genotyping microarrays using probe level information,
Komura D, Nishimura K, Ishikawa S, Panda B, Huang J, Nakamura H, Ihara S, Hirose M, Jones KW and Aburatani H,
In Silico Biology. 6, 72-92 2006. - Packet traffic analysis of scale-free networks for large-scale network-on-chip design,
Nobuhiko Oshida and Sigeo Ihara,
Physical Review E 74: (2) 026115-026122 August 2006. - Graph theoretical properties of protein interaction networks in the literature,
Sigeo Ihara, Rikako Suzuki, Junichi Kitakami, Naoko Nishikawa, Koji Abe, Shingo Tsuji, Shuichi Tsutsumi, Shogo Yamamoto, Michitaka Hirose, and Hiroyuki Aburatani,
Proceedings of the 17th International Symposium on Mathematical Theory of Networks and Systems, 1171-1121 2006. - Multidimensional support vector machines for visualization of gene expression data,
Komura D, Nakamura H, Tsutsumi S, Aburatani H, and Ihara S,
Bioinformatics vol. 21 issue 4 439-444 2005. - Analysis of mechanism for human γδ T cell recognition of non peptide antigens,
Seiji Yamashita, Yoshimasa Tanaka, Shuichi Tsutsumi, Hiroyuki Aburatani, Nagahiro Minato, Sigeo Ihara,
Biochemical and Biophysical Research Communications 334, Issue 2, 26 August 2005, Pages 349–360. - Interpreting expression profiles of cancers by genome-wide survey of breadth-of-expression in normal tissues,
X. Ge, Yamamoto S, Tsutsumi S, Midorikawa Y, Ihara S, S. M. Wang, and Aburatani H,
Genomics Volume 86, Issue 2, August 2005, Pages 127–141. - Protein interaction networks from literature mining,
Sigeo Ihara,
American Physical Society, APS March Meeting, March 21-25, 2005. - Identification of soluble NH2-terminal fragment of glypican-3 as a serological marker for early-stage hepatocellular carcinoma,
Hippo Y, Watanabe K, Watanabe A, Midorikawa Y, Yamamoto S, Ihara S, Tokita S, Iwanari H, Ito Y, Nakano K, Nezu J, Tsunoda H, Yoshino T, Ohizumi I, Tsuchiya M, Ohnishi S, Makuuchi M, Hamakubo T, Kodama T, Aburatani H,
Cancer Research 64, 2418 –2423, April 1, 2004. - Multidimensional support vector machines for visualization of gene expression data,
Komura D, Nakamura H, Tsutsumi S, Aburatani H and Ihara S,
Proceedings of 2004 ACM Symposium on Applied Computing, Nicosia, Cyprus 175-179 (2004). - Identification of gene expression profile in tolerizing murine cardiac allograft by co-stimulatory blockade,
Matsui Y, Saiura, A, Sugawara Y, Sata M, Naruse K, Yagita H, Kohro T, Mataki C, Izumi A, Yamaguchi T, Minami T, Sakihama T, Ihara S, Aburatani H, Hamakubo T, Kodama T, Makuuchi M,
Physiological Genomics 15: (3) 199-208 (2003). doi: 10.1152/physiolgenomics.00086.2003 - Cell simulation for circadian rhythm based on Michaelis-Menten model,
Sabau S.V, Hashimoto S, Nemoto Y, Ihara S,
Journal of Biological Physics 28: (3) 465-469 (2002). doi: 10.1023/A:1020341412380
並列計算機関連およびヘテロジニアス計算ソフトウェア
- Hot carrier induced degradation due to multi-phonon mechanism analyzed by lattice and device Monte Carlo coupled simulation,
Ho S, Ohkura Y, Maruizumi T, Joshi P, Nakamura N, Kubo S, Ihara S,
IEICE TRANSACTIONS on Electronics Vol.E86-C No.3 p.336-349 March 2003. - Agent-based middleware system applied to particle-mesh heterogeneous scientific simulations,
Shirun Ho, Sigeo Ihara and Satoshi Itoh, Shoichi Kubo, Masato Ikegawa, Nobuyuki Mise and Richard Dm Schlichting,
Proceeding of Real World Computing Symposium 2000, pp.43-48 (2000). - A New M-sequence based Parallel Random Number Generator with Reduced Correction,
Y. Yamamoto ,N. Naono and S. Ihara,
Ninth SIAM Conference on Parallel Processing for Scientific Computing March 22-24, 1999 San Antonio, Texas. - 「連成解析用ミドルウェアを用いた複合シミュレーションにおけるエージェント処理の性能評価」,
久保 昭一、何 希倫、井原 茂男、伊藤 智、リチャード・シュリックッティング、渡邊克博,
情報処理学会第60回全国大会講演論文集(1) pp.49-50 (1999). - A common software platform for heterogeneous scientific simulations,
何 希倫、井原 茂男、伊藤 智,
Joint Symposium on Parallel Processing 99: P.221 (1999). - Agent middleware for heterogenious scientific simulations,
Shirun Ho, Sigeo Ihara, and Richard Schlichting,
Workshop of Agent Based High Performance Computing, Autonomous agent 99, 1999. - 「異種シミュレーションを結合するエージェントミドルウェアのプロトタイプ開発」,
何 希倫、井原 茂男、伊藤 智,
第47期第2回分子動力学部門委員会、講演会資料、pp.1-12 (出版1998). - A common software structure for heterogeneous computing,
Shirun Ho, Sigeo Ihara, and Richard Schlichting,
Proceedings of Real World Computing symposium 98, pp.137-142, (1998). - 「異なる計算手法を結合したヘテロジニアスコンピューテジング」,
何 希倫、井原 茂男,
第47回応用力学連合講演会、講演予稿集、Japan NCTAM: pp187-88 (1998). - Agent middleware for heterogeneous scientific simulations,
Shirun Ho, Satoshi Itoh, Sigeo Ihara, and Richard Schlichting,
Proceedings of the 1998 ACM/IEEE conference on Supercomputing.
材料・半導体関連
- Quantum Monte Carlo study of silicon self-interstitials defects,
W.K. Leung, R.J. Needs, G. Rajagopal, S. Itoh and S. Ihara,
VLSI Design 13: (1-4) 229-235 Sp. Iss. SI (2001). doi: 10.1155/2001/83797 - Cluster ion implantation: a molecular dynamics study,
S. Ihara, S. Itoh and J. Greer,
Materials Science in Semiconductor Processing 3: 91-95. (2000). doi: 10.1016/S1369-8001(00)00012-3 - Calculations of silicon self-interstitial defects,
W. K. Leung, R. J. Needs, G. Rajagopal, S. Itoh and S. Ihara,
Physical Review Letters 83: 2351-2354 (1999). doi: 10.1103/PhysRevLett.83.2351 - Mechanisms of cluster implantation in silicon: A molecular-dynamics study,
S. Ihara, S. Itoh and J. Kitakami,
Physical Review B 58: 10736-10744 (1998) - Molecular dynamics in semiconductor physics,
S. Ihara and S. Itoh,
Computer Materials Science 10: 80-87 (1998). doi: 10.1016/S0927-0256(97)00087-6 - Monte Carlo study of Si-n-MOSFETs including the quantization of carriers,
Yasuyuki Ohkura, Shin'ichiro Kimura, Shrin Ho, Sigeo Ihara,
Solid-State Electronics 42: (11) 1997-2004 (1998). doi: 10.1016/S0038-1101(98)00164-6 - Numerical analysis of discharge voltage and light emission efficiency in AC-PDPs,
S Ho, S Ihara, M Shiiki, K Suzuki, A Yuhara, M Ishigaki, R Sato, N Kouchi, Y Hatano,
Proceedings of the Fifth International Display Workshops 479-482(1998). - Effects of the screening and mirror charges on electron mobility in the subthreshold region of Si MOSFETs,
Y Ohkura, S Kimura, S Ihara,
Solid-State Electronics 39: (9) 1371-1377 (1996). doi: 10.1016/0038-1101(96)00050-0 - Stability and vibrational spectra of toroidal isomers of C240,
K. Esfarjani, Y. Hashi, S. Itoh, S. Ihara and Y. Kawazoe,
Zeitschrift für Physik D Atoms, Molecules and Clusters 41: 73-76 (1997). doi: 10.1007/s004600050292 - Electronic properties of polygonal defects in graphitic carbon sheets,
Tamura R, Akagi K, Tsukada M, Itoh S, Ihara S,
Physical Review B 56: (3) 1404-1411 (1997). doi: 10.1103/PhysRevB.56.1404 - Electronic structure of C240 rings,
Y. Hashi, K. Esfarjani, S. Itoh, S. Ihara and Y. Kawazoe,
Transactions of the Materials Research Society of Japan 20: p486-489 (1996). - Order-N tight binding molecular dynamics: application to giant fullerenes,
S. Itoh, P. Ordejon, D.A. Drabold, R.M. Martin and S. Ihara,
Science Reports of the Research Institutes, Tohoku University. Ser. A, Physics, Chemistry and Metallurgy 41: 163-169 (1996). - Structure of polygonal defects in graphitic carbon sheets,
S. Ihara, S. Itoh, K. Akagi, R. Tamura and M. Tsukada,
Physical Review B 54: 14713-14719 (1996). doi: 10.1103/PhysRevB.54.14713 - Electronic structure of helically coiled carbon nanotubes: relation between the phason lines and energy band features,
K. Akagi, R. Tamura, M. Tsukada and S.Itoh and S. Ihara,
Physical Review B 53: 2114-2120 (1996). doi: 110.1103/PhysRevB.53.2114 - Electronic states of nanostructures of curved graphitic carbon cages,
M. Tsukada, K. Akagi, R. Tamura and S. Ihara,
Surface Review and Letters 3: 827-834 (1996). - Molecular dynamics study of toroidal and helically coiled forms of graphitic carbon,
S. Ihara and S. Itoh,
Surface Review and Letters 3: 827-834 (1996). - On coupling the drift-diffusion and Monte Carlo models for MOSFET simulation,
MB Patil, Y Ohkura, T Toyabe, S Ihara,
Solid-State Electronics 38: (4) 935-936 Apr (1995). doi: 10.1016/0038-1101(94)00206-U - Reverse short-channel effect of threshold voltage in LOCOS parasitic MOSFETs,
J. Tanaka, T. Toyabe, H. Matsuo, S. Ihara, H. Masuda and F. Otsuka,
Solid-State Electronics 38: 567-572 (1995). doi: 10.1016/0038-1101(94)00161-8 - Short-channel-effect-suppressed sub-0.1-µm grooved-gate MOSFET's with W gate,
Kimura S, Tanaka J, Noda H, Totabe T, Ihara S,
IEEE Transactions on Electron Devices 42: (1) 94-100 (1995). doi: 10.1109/16.370030 - Helically coiled and toroidal cage forms of graphitic carbon,
S. Ihara and S. Itoh,
Carbon 33: 931-939 (1995). doi: 10.1016/0008-6223(95)00022-6 - Electronic structure of helically coiled cage of graphitic carbon,
K. Akagi, R. Tamura, M. Tsukada, S. Itoh and S. Ihara,
Physical Review Letters 74: 2307-2310 (1995). doi: 10.1103/PhysRevLett.74.2307 - The electron mobility transition in n-GaAs heavily doped channels,
Y. Ohkura, H. Mizuta, I. Ohbu, O. Kagaya, K. Katayama and S. Ihara,
Semiconductor Science and Technology, Volume 9, Number 5S: 811-814 (1994). - Transmission matrix approach for electron transport in inversion layers,
M.B. Patil, Y. Okuyama, Y. Ohkura and S. Ihara,
Solid-State Electronics 37: 1359-1365 (1994). doi: 10.1016/0038-1101(94)90192-9 - Ab initio geometry and stability of a C120 torus,
C. Greer, S. Itoh and S. Ihara,
Chemical Physics Letters 222: 621-625 (1994). doi: 10.1016/0009-2614(94)00386-6 - Isomers of the toroidal forms of graphitic carbon,
S. Itoh and S. Ihara,
Physical Review B 49 May 15.: 13970-13974 (1994). doi: 10.1103/PhysRevB.49.13970 - Simulation of sub-0.1µm MOSFET's with completely suppressed short-channel effect,
J. Tanaka, T. Toyabe, S. Ihara, S. Kimura,H. Noda and K. Itoh,
IEEE Electron Device Letters 14: 396-399 (1993). doi: 10.1109/55.225591 - Toroidal forms of graphitic carbon. II. Elongated tori,
S. Itoh and S. Ihara,
Physical Review B 48: 8323-8328 (1993). doi: 10.1103/PhysRevB.48.8323 - Helically coiled cage forms of graphitic carbon,
S. Ihara, S. Itoh and J. Kitakami,
Physical Review B 48: 5643-5647 (1993). doi: 10.1103/PhysRevB.48.5643 - Toroidal forms of graphitic carbon,
S. Ihara, S. Itoh and J. Kitakami,
Physical Review B 47: 12908-12911 (1993). doi: 10.1103/PhysRevB.47.12908 - A Study of Quantum Monte Carlo Simulation for the Hydrogen Cluster Ions,
S. Itoh, S. Ihara,
Physics and Chemistry of Finite Systems: From Clusters to Crystals Volume 374 of the series NATO ASI Series pp 605-610 (1992) - Electronic structure of a dimer-adatom-stucking-fault model for the Si(111) 7x7 reconstructed surface,
S. Ihara, T. Uda and M. Hirao,
Applied Surface Science 60-61: 22-28 (1992) doi: 10.1016/0169-4332(92)90391-A - Ab initio molecular dynamics study of defects on the reconstructed Si(100) surfaces,
S. Ihara, S.L. Ho, T. Uda, M. Hirao,
Physical Review Letters 65: 1909-1912 (1990). doi: 10.1103/PhysRevLett.65.1909 - Large-scale elastic-plastic indentation simulations via nonequilibrium molecular dynamics,
W. G. Hoover, A. J. de Groot, C. G. Hoover, I. Stowers, T. Kawai, B. L. Holian, T. Boku, S. Ihara and J. Belak,
Physical Review A 42: 5844-5853 (1990) . doi: 10.1103/PhysRevA.42.5844 - Monte Carlo study of correlated diffusion in solids,
S. Ihara and K. Suzuki,
Journal of the Physical Society of Japan 55: 1695-1702 (1986). doi: 10.1143/JPSJ.55.1695 - Molecular dynamics study of Li3N,
S. Ihara and K. Suzuki,
Physics Letters A Volume 110: 265-268 (1985). doi: 10.1016/0375-9601(85)90095-7 - Molecular dynamics study of Ag3SI,
S. Ihara and K. Suzuki,
Journal of the Physical Society of Japan 54: 2607-2619 (1985). doi: 10.1143/JPSJ.54.2607 - Molecular dynamics study of β-AgI1_xClx
S. Ihara and K. Suzuki,
physica status solidi (b) 131: (1): 97-103 (1985). doi: 10.1002/pssb.2221310108 - Ionic conductivity in AgI1_xClx,
S. Ihara, Y. Warita, K. Suzuki,
physica status solidi (a) 86: 729-734 (1984). doi: 10.1002/pssa.2210860232 - Monte Carlo study of diffusion in non-Bravais lattice: application to the bcc lattice,
S. Ihara and K. Suzuki,
Journal of the Physical Society of Japan 53: 3476-3480 (1984). - Molecular dynamics study of α-Ag2S,
S. Ihara and K. Suzuki,
Journal of the Physical Society of Japan 53: 3081-3087 (1984). doi: 10.1143/JPSJ.53.3081
招待講演
- 転写の高時間分解時系列実験と数理解析:分子生物学の革新への期待, n-bioセミナー 長浜バイオ大学 2016/7/12.
- A Dynamical Study of Transcription in Human Cell, ‘RMT2015:from fundamental mathematics to biological applications’, OIST, Okinawa, 2015/11/6.
- Annulus diagram of SO(3) rotation of protein modules, ‘Géométrie et Biophysique’, CNRS; Institut de Recherche Mathématique Avancée, University of Strasbourg, France, 2015/9/19.
- A Diagram Representation of Interactions of Modules in Biological Molecules, 招待特別講演 IHÉS (Institut des Hautes Études Scientifiques), Bures-sur-Yvette France, 2015/9/14.
- iBMath Project: An integrative approach of biology and mathematics for dynamical transcription, ‘Geometry and topology of macromolecule folding’, Aarhus University, Denmark 2013/12/04.
- 転写過程のモデリングと動態シミュレーション, 応用数学連携フォーラム, 第8回生命科学者のための使える数学セミナー, 東北大学 星陵キャンパス 2013/7/12.
- Modeling and Simulation for Gene Transcription, ‘Moduli Spaces and Macromolecules’ IHÉS, Bures-sur-Yvette France, 2013/05/17.
- Protein interaction networks from literature mining, American Physical Society March Meeting, San Francisco, 2005/03/24.
- Molecular Dynamics in Semiconductor Research, The 10th Japan-Germany Forum on Information Technology 1996, Kroster-Seeon, Germany, Keynote Speaker, 1996/05/03.
- Toroidal and helical forms of graphitic carbon, 4th International Conference on Advanced Materials, Cancun, Mexico, 1995/08/27.
出版
- バイオテクノロジーの流れ(化学工業日報社、2002)共著 太田隆久 監修
- はじめての並列プログラミング(共立出版、1998)共著
- 先端材料の基礎知識(オーム社、1991)共著
新聞掲載
- 日本工業新聞 「文献検索による蛋白質相互作用ネットワーク構築」 平成15年11月21日 他5件
表彰
- 日経サイエンス主催 第3回コンピュータ ビジュアリゼーション コンテスト優秀賞受賞

